泸州老窖-江南大学国宝生态研究协同创新中心 毛健团队在国际期刊Briefings in Bioinformatics(Q1,IF=7.7)上发表了题为“GKNnet: an relational graph convolutional network-based method with knowledge-augmented activation layer for microbial structural variation detection(基于关系图卷积网络的知识增强激活层微生物结构变异检测方法)”的研究论文,重点比较了深度学习模型和传统检测方法相比的优越性,着重从精准率和召回率两方面进行研究开发,并根据F1分数的结果来验证了模型的优秀性能和稳定性。本论文获得国家自然科学基金(22422807,222478159)的资助。

文章背景
-
微生物基因组中的结构变异(SVs)在表型变化、环境适应以及物种进化中发挥着关键作用,尤其是缺失变异与表型性状密切相关。因此,准确且全面地识别缺失变异具有重要意义。尽管长读长测序技术能够检测到更多的结构变异,但其较高的错误率也引入了大量噪声,导致现有结构变异检测算法出现较高的假阳性率和较低的召回率。
-
本研究提出了一种基于图卷积网络(GCN)的结构变异检测方法。该方法首先通过构建异构图表示节点特征,并利用图卷积网络精确识别变异区域。同时,引入具有可学习激活函数的知识增强激活层(KANLayer),以降低变异区域周围的噪声,从而提升模型的精准率并减少假阳性。随后,通过聚类算法将多个重叠区域聚合为一个准确的变异区间,从而进一步提升召回率。
-
在模拟数据和真实数据集上的验证结果表明,该方法在F1分数上优于现有的基准方法(cuteSV、Sniffles、Svim 和 Pbsv),展现出其在结构变异检测中的优势与鲁棒性,为微生物基因组结构变异研究提供了一种创新的解决方案。
文章作者
研究亮点
(1)提出了一种专门针对于结构变异检测上下文的构图策略。
(2)将Kanlayer模块和MLP融合进行模型架构创新。
(3)在聚类算法上进行了筛选公式改进。
图文赏析
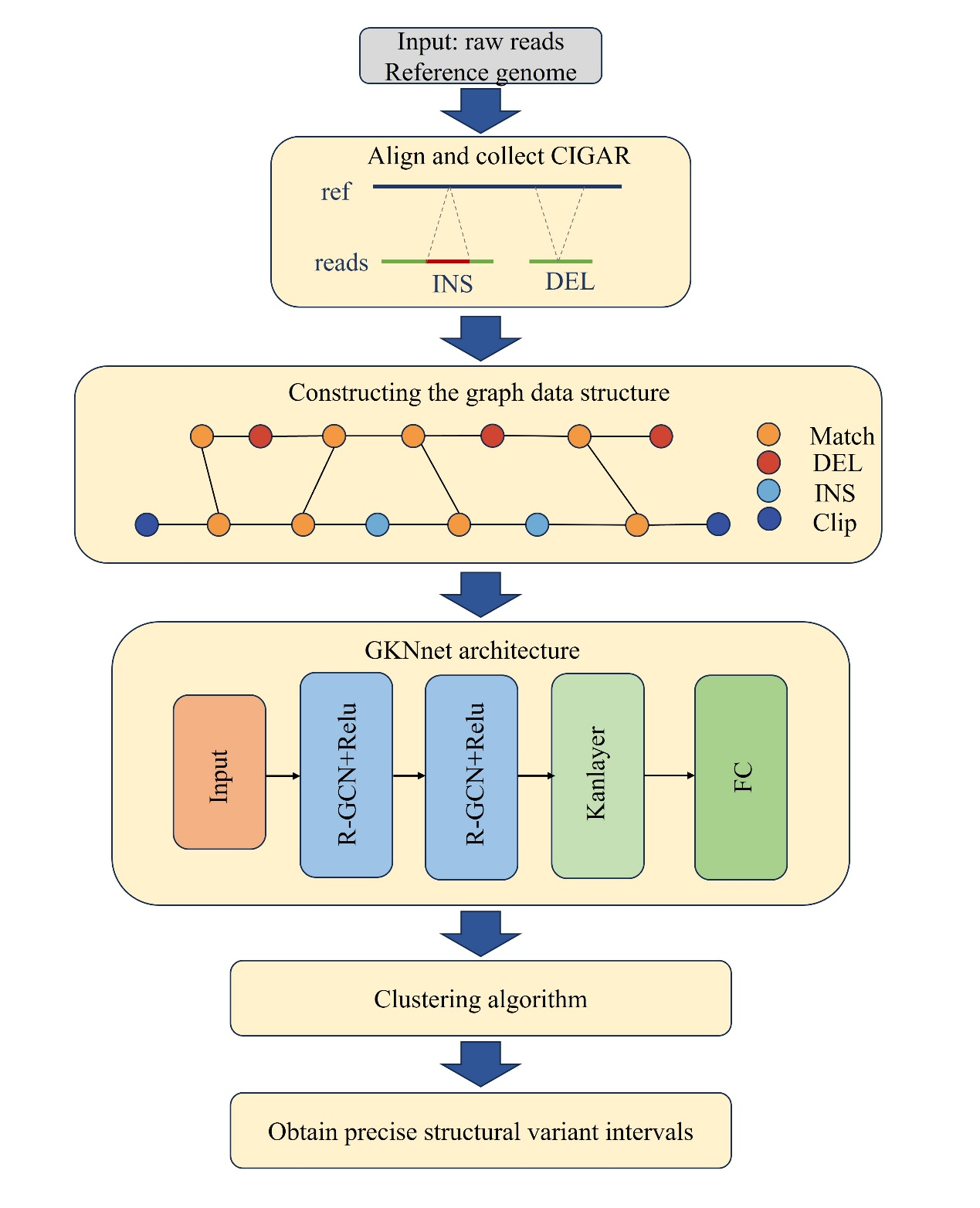
图1 数据流程图。(a)比对信息提取:将测序数据比对到参考基因组,提取 CIGAR 信息。(b)表征信息构建:通过图构建策略将 CIGAR 信息转换为图数据结构。(c)节点分类:使用 GKNnet 模型识别变异节点。(d)节点聚类:应用新的聚类策略以获得准确的结构变异(SVs)。
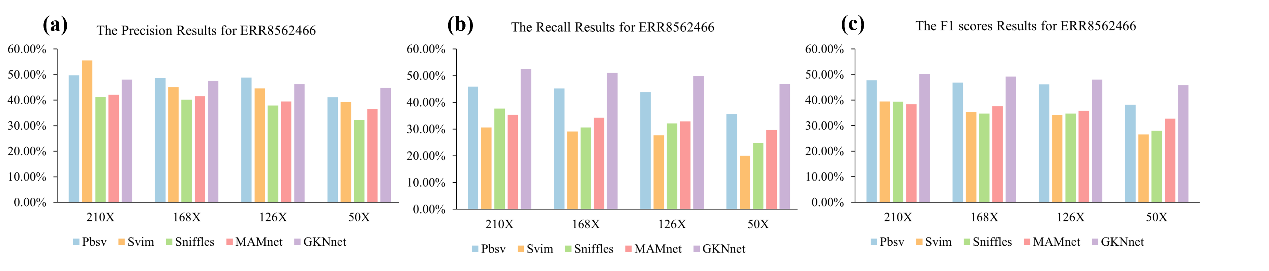
图2 (a)在 ERR8562466 数据集上进行缺失变异检测的精准率结果;(b)在 ERR8562466 数据集上进行缺失变异检测的召回率结果;(c)在 ERR8562466 数据集上进行缺失变异检测的 F1 分数结果。
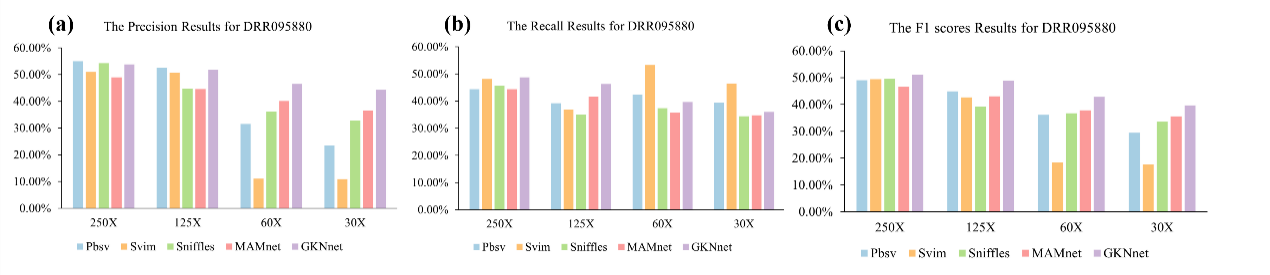
图3 (a)在 DRR095880 数据集上进行缺失变异检测的精准率结果;(b)在 DRR095880 数据集上进行缺失变异检测的召回率结果;(c)在 DRR095880 数据集上进行缺失变异检测的 F1 分数结果。
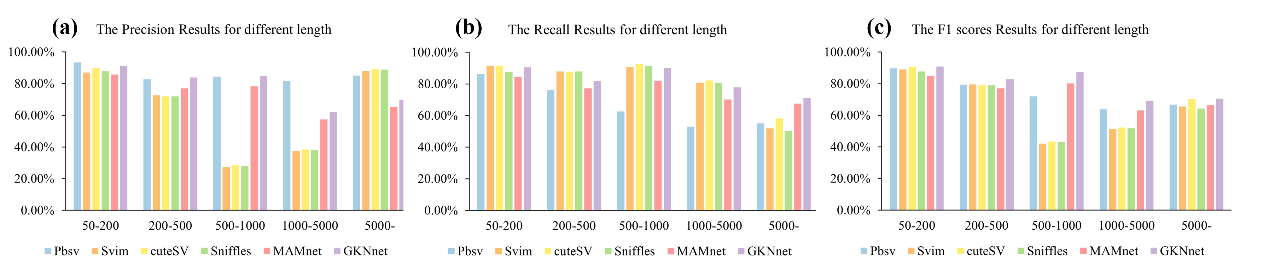
图4(a)在模拟数据集中,不同长度分组下各方法实验的精准率结果;(b)在模拟数据集中,不同长度分组下各方法实验的召回率结果;(c)在模拟数据集中,不同长度分组下各方法实验的 F1 分数结果。
原文链接:https://doi.org/10.1093/bib/bbaf200
